- Isolation and Characteristics of Escherichia coli from Groundwater in Agricultural Area
Dong-Hun Kim1*, Jung-Yun Lee2, Bong-Joo Lee1, and Byong Cho1
1Groundwater Environment Research Center, Geo-Environment Research Division, Korea Institute of Geoscience
and Mineral Resources, Daejeon 34132, Republic of Korea
2Environmental Biological Systems Research Institute, Chungnam National University, Daejeon 34134, Republic of Korea- 농업지역 지하수로부터 대장균 분리 및 특성 연구
김동훈1*ㆍ이정윤2ㆍ이봉주1ㆍ조병욱1
1한국지질자원연구원 지구환경연구본부 지하수환경연구센터, 2충남대학교 환경생물시스템연구소
This article is an open access article distributed under the terms of the Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/4.0) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.
Groundwater plays a crucial role in agricultural water supply in rural areas, but it is susceptible to contamination due to intensive agricultural activities and inadequate management. This study investigates the contamination status of Escherichia coli in groundwater from an agricultural region and characterizes the phylogroups and virulence factors of the isolated E. coli strains. A total of 140 E. coli strains were isolated from groundwater in the study area. Phylogenetically, Group A was the most abundant (47.9%), followed by Group B1 (25.0%), Group D (22.1%), and Group B2 (5.0%). Among the isolated E. coli strains, the detection frequency of major virulence genes was highest for stx2(35.0%), followed by ibeA (29.3%) and stx1 (12.9%). Pathotype analysis revealed that Shiga toxin-producing E. coli (STEC) accounted for 45.7%, and Neonatal Meningitis E. coli / Avian Pathogenic E. coli (NMEC/APEC) was also identified in 20.0%. These results suggest that groundwater in agricultural areas can be contaminated by various pathogenic E. coli strains, particularly STEC, which poses significant implications for groundwater hygiene management and public health. Therefore, continuous monitoring and the establishment of effective management systems for contamination sources are necessary for the safe and sustainable use of groundwater in agricultural areas.
Keywords: Groundwater, Agricultural area, Escherichia coli, Phylogenetic group, Virulence gene
물은 인간의 삶에 있어 가장 중요한 자원이지만 기후변화와 더불어 인구 증가, 산업화, 도시화에 따른 물 수요가 증가함에 따라 그 중요성이 더욱 부각되고 있다(Jha et al., 2020). 특히, 지하수는 전 세계적으로 인간이 사용할 수 있는 가장 풍부하고 중요한 물 공급원으로 인식되어 왔다(Abia et al., 2017).
농어촌지역을 포함한 전국의 상수도 보급률은 지난 10년간 꾸준히 증가하여 2023년 기준 99.5%에 해당하며, 연구지역의 경우 마을 상수도 및 소규모 급수시설을 포함한 상수도 보급률이 95.1%에 이른다(KOSIS, 2025). 하지만, 농업지역의 경우, 농업·공업·생활용수의 대부분을 지하수에 의존하고 있어 지하수의 이용률이 높아 이에 맞는 효과적인 지하수 관리가 필수적이다(Yoon et al., 2020). 특히, 농업지역에서는 충적층과 암반풍화대에 존재하는 천부 지하수를 주로 사용하는 것으로 알려져 있어 강우 등 인근 지표수의 함양은 지하수 충진과 동시에 지하수 오염을 유발할 수 있다(Boulding and Ginn, 2016; Kim et al., 2022). 이러한 농업지역 지하수 오염원으로는 화학비료, 시비, 축산폐수 유출 등의 농업활동과 관련이 높으며 정화조 및 하수처리 시설로부터 유출되는 경우도 있다(Lee and Murphy, 2020).
농업지역의 지하수 오염은 전 세계적인 문제로 대두되고 있으며, 작물 관개 자원으로서의 지하수 이용 가능성을 제한하고 있다. 특히 관개 용수 관련 식중독과 같은 수인성 질환의 발생은 인간의 건강문제 뿐만 아니라 농업 소득 손실을 초래할 수 있다(Bartzas et al., 2015). 오염된 지하수의 음용 및 관개 용수로의 사용과 관련된 식중독과 같은 수인성 질환은 지속적으로 발생하고 있다. 대표적인 감염 사례로 2006년 미국 캘리포니아에서 농업용수로 세척된 포장된 시금치를 섭취하고 대규모로 발생한 장출혈성 대장균 감염(E. coli O157:H7)(Gelting et al., 2011), 로메인 상추와 잎채소를 매개로 한 식중독(Viazis et al., 2025; Yang et al., 2025), 신선 토마토 관련 여러 주에 거쳐 발생한 감염(Pagadala et al., 2015), 혼합된 샐러드를 통해 영국에서 발생한 대규모 감염(Gobin et al., 2018) 사례 등이 있다. 최근 국내에서도 학교 캠프 중 음용수 관련 다발성 설사원성 대장균 감염이 집단으로 발생하기도 하였다(Park et al., 2018).
분변 지표균인 대장균에 의한 지하수 오염은 잠재적인 분변 오염 및 기타 유해 병원균의 존재 가능성을 시사한다. 이러한 지하수의 대장균 오염은 가축 분뇨, 정화조의 부적절한 처리, 지표수와의 연결 등에 의해 발생한다(Masters et al., 2011). 일반적으로 대장균은 인간을 포함한 온혈동물의 장에서 발견되는 공생균으로 여겨지며, 대부분의 장내 대장균은 비병원성 공생균이지만, 일부 균주는 병원성 유전자를 보유하여 설사나 용혈성 대장염과 같은 장 감염을 유발하거나 신생아 수막염, 병원내 패혈증, 용혈성 요독 증후군과 같은 장외 감염을 유발할 수도 있다(Kaper et al., 2004; Masters et al., 2011). 대장균은 계통분류학적으로 A, B1, B2, D 그룹을 포함한 다양한 계통군으로 분류될 수 있으며, 각 그룹에 속하는 균주는 서로 다른 표현형 및 유전형 특성을 갖고 생태적 서식지 또한 다른 특징을 갖는다(Clermont et al., 2013; Jang et al., 2014; Stoppe et al., 2017). 따라서, 대장균 균주에 대한 계통학적 동정은 해당 균주의 생리학적 및 생태학적 측면에서 중요한 정보를 제공할 수 있다.
대장균 중에는 특정 병원성 인자를 획득하여 새로운 환경에 적응하는 능력을 향상시키고 광범위한 질병을 유발할 수 있도록 적응된 균주가 존재한다. 하지만, 단일 또는 다중의 병원성 유전자를 보유한다고 해서 해당 균주가 반드시 병원성을 나타내는 것은 아니며, 적절한 병원성 인자의 조합을 보유해야 한다(Gilmore and Ferretti, 2003; Masters et al., 2011). 이러한 병원성 인자의 조합을 통해 질병을 유발할 수 있는 대장균에 감염되면 장/설사 질환, 요로 감염, 패혈증/수막염과 같은 임상 증후군이 발생할 수 있다. 대장균은 병원성 인자, 감염 기전, 장세포와의 상호 작용, 조직 친화성, 증상 및 증후군에 따라 다양한 병원형으로 분류된다(Geurtsen et al., 2022; Kaper et al., 2004). 대장균의 병원형은 질병의 발생 위치에 따라 크게 장내 병원성 대장균(InPEC, Intestinal Pathogenic E. coli)과 장외 병원성 대장균(ExPEC, Extraintestinal Pathogenic E. coli)의 두 그룹으로 구분된다. 장내 병원성 대장균은 i) 사람에게 심각한 출혈성 대장염을 유발하며 병원균 중 가장 널리 알려진 시가 독소 생성 대장균(STEC, shiga toxin producing E. coli), ii) STEC의 한 유형으로 출혈성 대장염과 용혈성 요독 증후군(hemolytic-uremic syndrome, UHS)을 유발하는 장출혈성 대장균(EHEC, enterohaemorrhagic E. coli), iii) 여행자 설사를 유발하는 장독소원성 대장균(ETEC, enterotoxigenic E. coli), iv) 동물과 소아 모두에서 설사를 유발하는 부착 및 소실 장 병변 형성을 특징으로 하는 장병원성 대장균(EPEC, enteropathogenic E. coli), v) 이질을 유발하는 장침습성 대장균(EIEC, enteroinvasive E. coli), vi) 인간에게 지속적인 설사를 유발하는 장응집성 대장균(EAEC, enteroaggregative E. coli)과 vii) 소아에게 설사를 유발하는 확산부착성 대장균(DAEC, diffusely adherent E. coli)으로 세분할 수 있다(Denamur et al., 2021; Kaper et al., 2004; Mageiros et al., 2021; Masters et al., 2011; Rojas-Lopez et al., 2018). 장외 감염성 대장균은 요로병원성 대장균(UPEC, uropathogenic E. coli), 신생아 수막염 대장균(MNEC, meningitis neonatal E. coli), 패혈증 관련 대장균(SEPEC, sepsis-associated E. coli)과 조류 병원성 대장균(APEC, avian pathogenic E. coli)으로 분류된다(Denamur et al., 2021; Geurtsen et al., 2022; Sarowska et al., 2019).
대장균으로 인한 식품 매개 또는 수인성 질병은 전 세계적인 공중 보건 문제로 인식되고 있기 때문에 이러한 감염원을 파악하고 치료할 수 있도록 대장균을 빠르고 효과적이며 정확하게 검출하는 것이 매우 중요하다(Alhadlaq et al., 2024; Majowicz et al., 2014). 병원성 대장균의 검출 및 동정을 위해 선택적 배양법, 면역진단 검사 방법, 광학적 기술, 분자생물학적 방법, 전기화학적 방법 등 다양한 기술이 사용되어 왔다. 최근 미생물 유전체 분석 기술의 발달로 광범위한 유전자 정보를 통해 병원성 대장균 특이적 유전자 정보를 확보 가능함에 따라 신속 정확하고 비용 효율적인 진단 검사 기법이 활발히 개발 중이다(Abbas et al., 2023; Govindarajan et al., 2024; Xue and Zhang, 2025).
지하수 의존도가 높은 농업지역에서 지하수의 오염은 농산물의 안전성 저하로 인한 농업 소득 손실뿐만 아니라 지하수를 이용하는 주민의 건강에도 직접적인 위험이 될 수 있다. 하지만 국내의 경우 농업지역 지하수의 유해미생물 오염에 대한 관리가 미흡한 실정이다. 본 연구에서는 농업용수 및 생활용수로 사용되는 노성천 유역 지하수에서 분리한 대장균의 계통분류학적 및 병원성 유전자 존재 유무에 대한 연구를 수행함으로써, 노성천 유역 지하수의 잠재적 위험성을 평가하였다.
2.1. 연구지역 및 시료 채취
연구지역인 노성천 유역은 전형적인 농촌지역으로 충청남도 공주시와 논산시를 지나는 길이 27.7 km, 유역 면적은 202.5 km2인 노성천을 중심으로 농업활동이 활발히 이루어지고 있다. 연구지역의 연평균 기온과 강수량은 각각 12.2°C와 1349.2 mm이고, 최근 10 년간 논산시의 지하수 사용량은 연평균 29.5백만m3로 점차 증가하는 경향을 보이고 있다(Kim et al., 2022). 특히, 농업용수로의 이용이 생활용수와 공업용수의 사용량에 비해 2.4배 이상으로 대부분의 지하수가 농업활동에 사용되며 이러한 농업용수의 비중이 점차 증가하고 있다(충청남도 지하수 통합정보시스템, 2025). 이는 하류 지역에서 집중적으로 이루어지는 지하수를 이용한 수막재배의 영향으로 판단되며, 과도한 지하수 이용으로 인해 심각한 지하수위 하강을 유발하여 하천 유실과 지하수 온도 감소 문제를 발생시키기도 한다(Cho et al., 2012; Kwon et al., 2020).
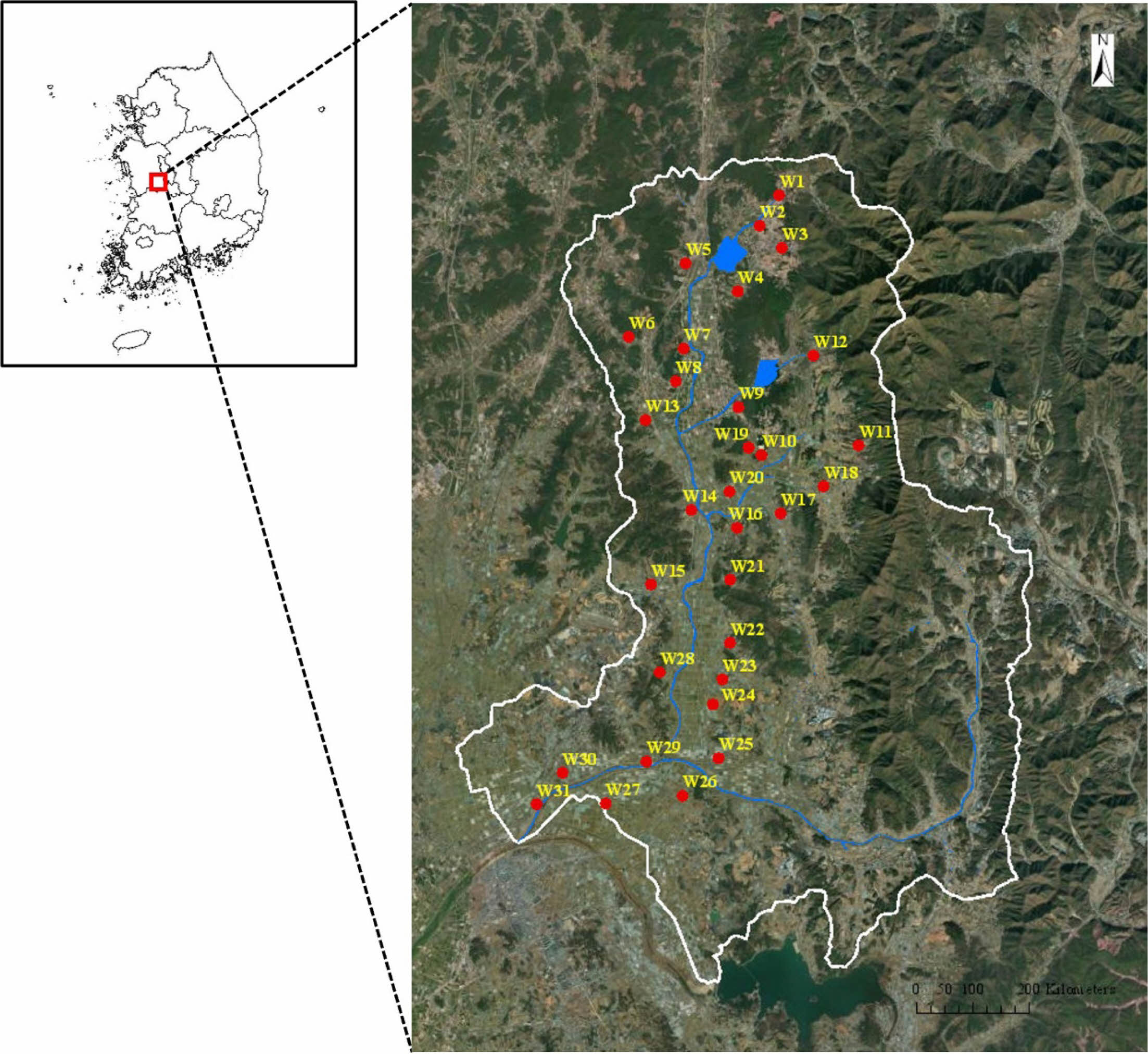
본 연구에 사용된 지하수 시료는 2018년 9월에 노성천 유역에 위치한 31개의 개인 또는 공용 지하수 관정에서 채취하였다(Fig. 1). 해당 관정의 심도는 20 – 1,000 m로 다양하였으나, 대부분 30 m 이내의 천부 관정이었고 관정 주변은 주로 농지로 이루어져 있었다. 또한, 눈에 띄는 지표 오염원은 발견되지 않았다. 지하수의 현장 수질(수온, 전기전도도, pH, 용존 산소, 산화환원전위)은 휴대용 수질측정장비(Multi 3620 IDS, WTW, Germany)를 이용하여 시료 채취 직전에 측정하였으며, 시료는 현장 수질이 안정될 때까지 지하수를 purging 한 뒤 채취하였다. 지하수 중 주요 용존 양이온(Ca2+, Mg2+, Na+, K+, SiO2 (aq))과 용존 음이온(Cl-, SO42-, NO3-, HCO3-) 분석을 위하여 현장에서 0.45 μm membrane filter (Advantec, Japan)를 이용하여 부유물질을 제거한 후 각각 30 mL를 폴리에틸렌 용기에 채취하였다. 용존 양이온 시료는 용기 벽면 흡착과 침전을 방지하기 위해 현장에서 질산(HNO3)을 첨가하여 pH를 2 이하로 유지하였다. DOC (Dissolved Organic Carbon) 시료는 20 mL 갈색 유리용기에 채취하였으며 H2SO4를 사용하여 산처리하였다. 대장균 분리를 위해 시료 1 L를 무균 채수병에 채취하여 냉장 상태(4°C)를 유지하며 실험실로 운반하였으며, 12시간 이내에 대장균 분리를 위한 배양을 수행하였다.
2.2. 주요 용존 이온 분석
지하수 시료의 주요 용존 양이온과 음이온 농도는 각각 Inductively coupled plasma-optical emission spectrometry (ICP-OES; Optima 7300 DV; Perkin Elmer, USA)와 Ion-exchange chromatography with conductivity detection (IC; ICS-1500; Dionex, USA)를 이용하여 측정하였다. HCO3 (total alkalinity) 농도는 총 알칼리도 측정 시약(TNT 870 kit; Hach, USA)을 첨가하여 나타나는 발색반응을 분광 광도계(DR-1500, Hach, USA)를 이용하여 측정하였다. DOC는 Total organic carbon analyzer (TOC-VCPH; Shimadzu, Japan)를 하여 측정하였으며 측정 한계는 0.002 mg/L이다. Ca2+, Mg2+, Na+, K+, SiO2 (aq), Cl-, SO42-, NO3- 농도의 측정한계는 각각 0.02, 0.1, 0.1, 0.01, 0.06, 0.01, 0.05, 0.04 mg/L이다.
2.3. 대장균 분리
대장균은 지하수 시료로부터 막 여과 기술을 이용하여 분리하였다(USEPA, 2014). 시료 1 L를 47 mm 크기의 0.45 μm 멤브레인 필터(Advantec, Japan)를 사용하여 여과하였고, 필터를 m-TEC 고체 배지(Difco, USA) 위에 올려놓고 35oC에서 2시간 배양 후 44.5oC에서 16시간 배양하였다. 적색 또는 자홍색을 나타내는 집락은 새로운 m-TEC 고체 배지에 다시 배양하여 단일 집락으로 분리하였다. 잠재적인 대장균 분리 균주는 다른 대장균군이나 그람음성균과 구분되어 파란색 집락을 형성하는 ChromAgar ECC (Chromagar Microbiology, France)에 배양시켜 대장균임을 재확인하였다(Dombek et al., 2000; Jang et al., 2011; Unno et al., 2009). 분리된 대장균 균주는 다음 실험에 사용될 때 까지 LB (Luria-Bertani) 냉동 배지(Green and Sambrook, 2012)로 옮겨 -70oC에 보관하였다.
2.4. DNA 추출 및 PCR 반응
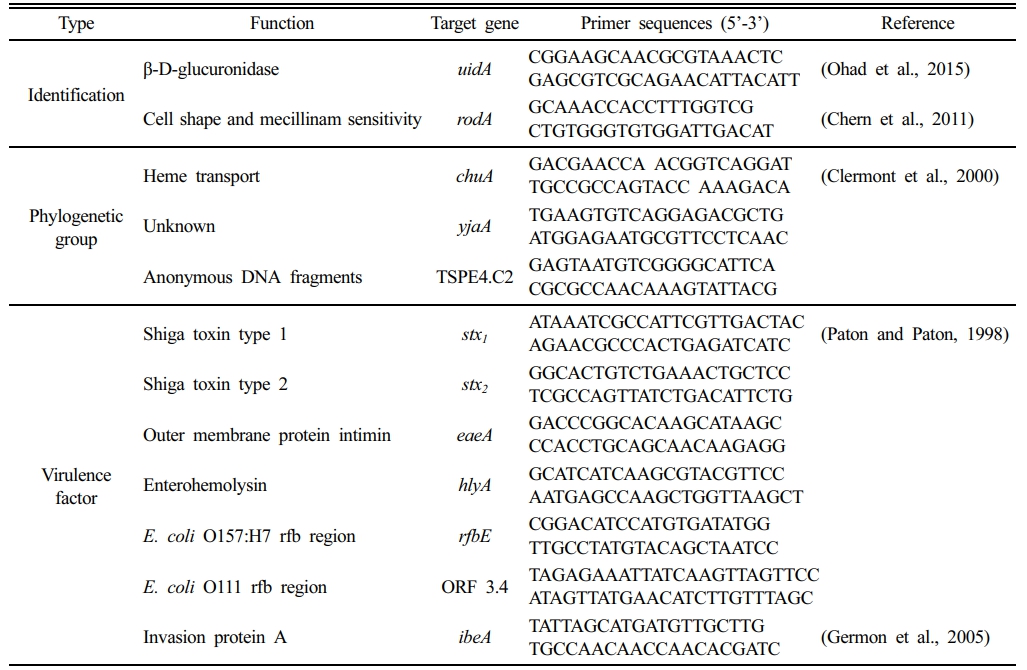
분리된 대장균 균주들의 gDNA 추출을 위하여 각 균주들을 LB 배지 1 mL로 채워진 deep well plate에서 37oC에서 12시간 배양한 뒤 Genomic DNA extraction kit (Bioneer Co., Korea)를 사용하여 추출하였다. 추출된 gDNA를 주형으로 대장균 여부 확인, 계통분류학적 분류 및 병원성 유전자 존재 여부를 검출하는 PCR 반응을 각각 수행하였다(Table 1). 분리된 균주들의 대장균 여부를 확인하기 위하여 대부분의 대장균에서 발견되는 복합 탄수화물의 가수분해하는 효소인 β-glucuronidase를 코딩하는 uidA 유전자와 세포 모양과 mecillinam 민감도에 영향을 미치는 불특정 단백질을 코딩하는 rodA 유전자를 대장균 특이적 마커 유전자로 선정하여 PCR을 수행하였다(Chern et al., 2011; Martins et al., 1993; Ohad et al., 2015). 대장균의 계통분류학적 분류는 heme 수송에 필요한 chuA 유전자와 기능이 알려져 있지 않은 yjaA 유전자, 유전체 라이브러리에서 TSPE4.C2로 지정된 DNA 단편을 대상으로 다중 PCR 방법을 사용하여 수행하였다(Clermont et al., 2000; Escobar-Páramo et al., 2006; Jang et al., 2014). 또한, 대장균 균주들 중 시가 독소 생성 대장균(STEC)과 Shigella dysenteriae 균주에서 생성되는 강력한 세균성 독소인 시가 독소(Shiga toxin) 생성 관련 유전자(stx1과 stx2) (Paton and Paton, 2002; Shimizu et al., 2009), 장병원성(EPEC) 및 장출혈성 대장균(EHEC)에 의해 유발되는 부착-소실 병변에 필수적인 세균 부착소인 intimin 관련 유전자(eaeA) (Agin and Wolf, 1997), 장출혈성 대장균에서 생성되는 강력한 독력인자 enterohemolysin 관련 유전자(hlyA) (Kaper et al., 2004), 식품 매개 장출혈성 대장균의 대표적 혈청형으로 혈변, 복통, 용혈성 요독 증후군(HUS)과 관련이 있는 serogroup O111과 O157을 대상으로 하는 유전자(rfbEO157:H7, ORF 3.4) (Bastin and Reeves, 1995; Bilge et al., 1996), 신생아 수막염 유발 대장균(NMEC) 및 조류 병원성 대장균(APEC)에서 병원성 인자로 발견되는 유전자(ibeA) (Germon et al., 2005; Paton and Paton, 1998; Wang et al., 2011)의 존재 여부를 확인하였다.
PCR 반응은 EmeraldAMP® PCR Master Mix (Takara Co., Ltd., Japan)를 사용하였으며, PCR 반응액은 25 μl의 Master Mix에 1 μl의 주형 DNA, 각 1 μl (10 pmole)의 primer, 나머지는 멸균 증류수를 첨가하여 최종 부피가 50 μl가 되게 하였다. 모든 PCR 반응은 Thermal Cycler Dice Real Time System Lite (TP700; Takara co., Ltd., Japan)를 사용하였으며 반응 조건은 기존 문헌을 참고하였다(Germon et al., 2005; Paton and Paton, 1998). PCR로 증폭된 생산물은 자동 모세관 전기영동 시스템인 Fragment Analyzer (5200; Agilent, USA)를 이용하여 확인하였다.
|
Fig. 1 Sampling locations of groundwater wells in the study area (Noseong stream watershed). |
|
Table 1 Primer sequences used for identification, phylogenetic group, and virulent profile analyses of E. coli isolates |
3.1. 지하수 수질 특성
기본적인 지하수 특성 조사를 위해 지하수의 현장 수질을 측정하였다(Table 2). 연구지역 지하수 수온은 14.2 ‒ 37.8oC 범위로 평균 17.6oC로 나타났고, pH는 5.7 ‒ 8.4의 범위로 대체로 일정하게 나타났으며 평균 6.5로 약산성의 특징을 보였다. 전기전도도(EC)는 65.7 ‒ 733 μS/cm 범위의 값을 갖으며 평균 247.9 μS/cm를 보였다. 용존 산소와 산화환원전위의 경우, 각각 0.2 ‒ 9.2 mg/L와 -124.7 ‒ 565.2 mV의 광범위한 범위를 보였다. 이는 연구지역은 전형적인 농촌 지역으로 조사된 관정들이 대부분 30 m 이내의 천부관정으로 주로 농업용과 생활용으로 사용되고 있어 지하수 사용량이 많아 주변의 물과 공기와의 접촉에 의한 영향으로 판단된다(Kim et al., 2022). 반면, W1 관정의 경우 심부 관정으로 온천수 활용을 위해 개발하였으나 현재는 농업용으로 사용하고 있어 높은 수온과 pH 값, 낮은 용존 산소와 산화환원 전위 값을 보였다. 전체적인 양이온 농도는 Ca2+ > Na+ > Mg2+ > K+ 순으로 나타났고, 음이온은 HCO3- > NO3- > Cl- > SO42- 순으로 지하수에 존재하는 것으로 나타났다. 양이온은 대부분 지질 기원에 의한 것으로 보이나 음이온과의 상관관계를 통해 농업활동에 의한 오염원 유입의 영향으로 비교적 높은 농도로 용존 되어 있는 것으로 판단된다(Kim et al., 2022). 음이온의 경우 NO3-와 Cl- 이온이 자연상태의 지하수 농도를 초과하고 있는 것으로 보아 인위적 오염의 유입을 지시하며 이는 전형적인 농촌지역의 농업활동과 더불어 생활 활동에 의해 지하수 오염이 진행되고 있는 것으로 판단된다(Mueller and Helsel, 1996).
TDS (Total Dissolved Solids)의 경우 용존 이온의 영향으로 41.9 – 456 mg/L의 광범위한 범위를 갖으며 평균 159.8 mg/L를 보였다. 연구지역의 DOC는 평균 2.1 mg/L로 0.9 – 4.2 mg/L의 범위를 갖는다. DOC는 주로 토양층을 통한 지표수의 유입에 의해 생성되지만 농업지역의 집중적인 토지 사용에 따른 유기물의 인위적 유입이 직접적인 원인이 되기도 한다. 이러한 DOC는 지하수 내 미생물의 생존 및 군집 구조와 밀접한 연관이 있으므로 수자원 관리 차원에서 관심이 요구된다(Bagordo et al., 2024; Li et al., 2012).
대장균의 경우 관정당 평균 4.5 균주가 분리되었으며 이는 연구지역 지하수의 분변 오염 가능성을 시사하지만 수질 인자와의 상관성에서 유의성을 보이지 않고 관정별로 분리된 대장균의 범위(0 ‒ 20)가 넓어 향후 수질과의 연관성에 대한 연구가 더 필요할 것으로 판단된다(Jang et al., 2017; Odonkor and Mahami, 2020).
3.2. 지하수의 대장균 분리
노성천 유역 31개 지하수 관정 시료로부터 두번의 선택배지를 이용하여 총 142개의 잠재적 대장균 균주를 분리하였다. 이들 중 대장균 특이적인 uidA와 rodA 유전자의 유무를 PCR 반응으로 판단하여 최종 140개의 균주를 선별하였다. 각 관정에서 분리된 대장균은 최소 0에서 최대 20 균주로 관정별로 다양하게 분포하였다(Fig. 2). 전체 31개 관정 중 58.1%에 해당하는 18개 관정에서 대장균이 분리되었다(Table 3). 관정의 유형에 따라 개인용 관정의 60%와 공용 관정의 54.5%에서 대장균이 분리되었다. 관정의 용도에 따라서는 농업용으로 사용하는 관정의 66.7%와 생활용 관정 52.6%에서 대장균이 분리되었다. 전체적으로 개인용 관정 중 생활용으로 사용하는 관정에서 가장 많은 대장균(50.7%)이 분리되었다. 대장균은 분변 오염을 나타내는 가장 적합한 지표로 간주되며, 일반적으로 사람과 동물의 배설물뿐만 아니라 하수에서도 대량으로 발생한다. 따라서 수질 시료에서 대장균의 검출은 분변 물질이 존재하고 유해한 질병을 유발할 수 있는 병원균이 존재할 가능성을 나타낸다(Gwimbi et al., 2019; Mutileni et al., 2023). 특히, 농촌지역에서 사용하는 개인용 관정의 경우 관리가 잘 이루어지지 않고 경작지와 인접해 있으며 생활용으로 사용하기 위해 가구 내에 관정이 위치하는 경우가 많아 국소적 오염에 더욱 취약할 것으로 판단된다. 또한, 농작물 재배를 위해 이용되는 농업용수에서 대장균이 검출되는 경우 농작물에 대한 안전성도 보장할 수 없을 뿐 아니라, 농가에 심각한 피해를 야기할 수도 있다(Holvoet et al., 2014; Jay et al., 2007; Tousi et al., 2021).
3.3. 분리 대장균의 계통분류학적 특성
대장균은 주로 A, B1, B2 및 D의 네 가지 주요 계통분류학적 그룹으로 분류할 수 있으며(Clermont et al., 2000), 최근 대장균 균주에 대한 광범위한 다중 유전자 서열 타이핑(Multi locus sequence typing, MLST) 및 유전체 데이터를 통해 주요 계통분류학적 그룹보다 비교적 드물게 발생하는 C, E, F 및 Escherichia clade I과 같은 새로운 계통분류학적 그룹에 대한 연구도 수행되고 있다(Clermont et al., 2013). 각 계통분류학적 그룹에 속하는 균주는 서로 다른 표현형 및 유전형 특성을 가지고 있으며 이들 네 가지 그룹은 생태적 서식지 또한 다르다고 보고되었다(Jang et al., 2014; Stoppe et al., 2017).
연구지역 지하수 시료로부터 분리된 대장균을 계통분류학적으로 구분한 결과 A, B1, B2, D 그룹이 각각 47.9, 25.0, 5.0, 22.1%로 분류되었다(Fig. 3). A 그룹이 전체 대장균의 대부분을 차지하였으며, B1과 D 그룹이 유사하게 존재하였고, B2가 가장 적게 존재하였다. 일반적으로 환경에서 A 또는 B1 그룹이 B2와 D 그룹보다 더 자주 발견되는 특징을 보인다(Jang et al., 2014). 관정 유형에 따라 개인용 관정에서는 A 그룹이 49.5%로 가장 높은 비율로 존재하였으며 B1 그룹이 18.8%, B2 그룹이 6.9%, D 그룹이 24.8%로 존재하고 있었다. 공용 관정에서는 A, B1, B2, D 그룹이 각각 43.6, 41.0, 0.0, 15.4%로 분포하고 있었다. 비록, 관정 유형별로 분리된 대장균의 수가 다르지만 A 그룹이 대장균의 계통분류학적 그룹의 대부분을 차지하고 있었으며 공용 관정에서 A 그룹과 B1 그룹이 유사한 비율로 분포하는 것으로 나타났다. 또한 공용 관정에서는 B2 그룹이 존재하지 않는 것으로 나타났다. 대장균의 계통군은 오염원인 숙주와 강한 상관관계를 보이기 때문에 계통군 분포와 오염원과의 연관성 파악을 위한 오염원 규명 연구가 추가적으로 수행될 필요가 있다.
관정 용도에 따라서는 개인 농업용 관정에서 A 그룹이 46.7%로 가장 높은 비율로 나타났고 B1, B2, D 그룹이 각각 20.0, 10.0, 23.3% 비율로 존재하고 있었다. 개인 생활용 관정에서는 50.7%가 A그룹으로 구분되었으며 D 그룹이 25.4%, B1과 B2가 18.3%와 5.6%로 나타났다. 공공 농업용 관정에서는 A, B1, B2, D 그룹에 각각 27.6, 51.7, 0.0, 20.7%를 차지하고 있었으며, 생활용 관정은 A 그룹과 B1 그룹이 각각 90.0%와 10.0%로 B2와 D 그룹은 존재하지 않는 것으로 나타났다. 대장균의 계통분류학적 분류는 집단 유전학, 장외 병원균 및 공생 균주의 분류, 숙주와 근원 관계 등의 연구에 유용하다. 인간의 장내에 공생하는 대장균은 대부분 A 그룹에 속하며 동물에서 분리한 균주는 대부분 B1 그룹에 속한다고 알려져 있다(Stoppe et al., 2017). 특히 개인용 관정의 경우 B2 그룹이 6.9%를 차지하는 것으로 나타났는데, B2 그룹은 다른 그룹에 비해 장외 감염을 유발하는 병원균의 경향이 더 높은 것으로 알려져 있다(Nowrouzian et al., 2009; Unno et al., 2009).
전제적으로 개인용 관정에서 용도와 관계없이 다양한 계통분류학적 그룹의 대장균이 분리되었으며, 공용 관정의 경우 농업용에서 더 다양한 그룹이 발견되었다. 따라서, 대장균 균주에 대한 계통분류학적 그룹 동정은 해당 균주의 생리학적 및 생태학적 측면에 대한 중요한 정보를 제공할 수 있다.
3.4. 분리 대장균의 병원성 유전자 분포
대장균은 일반적으로 출생 후 몇 시간 이내에 인간 유아의 위장관에 서식하며 숙주인 인간과 수 십년 동안 건강하게 공존하며 상호 이익을 추구하는 비병원성 공생 균주로 여겨진다(Geurtsen et al., 2022; Kaper et al., 2004; Masters et al., 2011). 하지만, 특정 균주의 경우 설사나 출혈성 대장염(hemorrhagic colitis)과 같은 장 감염을 유발하거나 신생아 수막염(neonatal meningitis), 병원감염 패혈증(nosocomial septicemia), 용혈성 요독 증후군(hemolytic uremic syndrome, HUS), 요로 및 수술 부위 감염과 같은 장외 감염을 유발할 수 있는 병원성 유전자 조합을 보유할 수도 있다(Masters et al., 2011). 따라서 본 연구에서는 연구지역 지하수에서 분리된 전체 대장균 140 균주를 대상으로 병원성 유전자 존재 여부를 확인하였다.
전체 140 균주 중 93 (66.4%) 균주에서 적어도 하나 이상의 병원성 유전자를 갖고 있는 것으로 나타났으며, 나머지 47 (33.6%) 균주는 병원성 유전자를 갖지 않는 것으로 나타났다(Table 4). 개인용 관정의 경우 전체 101개의 분리 대장균 중 64 (63.4%) 균주에서 하나 이상의 병원성 유전자가 검출되었으며, 공공 관정에서는 전체 39 분리 균주 중 29 (74.4%) 균주에서 병원성 유전자가 검출되었다. 지하수 용도에 따라서는 개인 관정의 경우 농업용과 생활용 모두 분리 균주의 60% 이상에서 병원성 유전자가 검출되었으며, 공공 관정에서는 두 용도 모두에서 분리 균주의 70% 이상이 병원성 유전자를 갖는 것으로 나타났다.
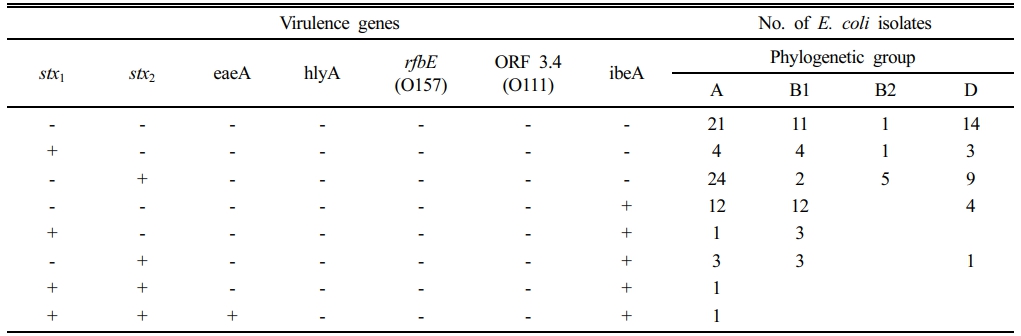
병원성 유전자 하나를 포함하는 균주는 전체 분리 대장균 중 57.1% (n = 80)에 해당하였으며, A 그룹이 28.6% (n = 40), B1 그룹 12.9% (n = 18), B2 그룹 4.3% (n = 6), D 그룹 11.4% (n = 16)으로 분포되어 있었다(Fig. 4, Table 4). 병원성 유전자는 stx2> ibeA> stx1 순으로 검출되었으며, 서로 다른 병원성 유전자 두 개를 포함하는 균주는 전체의 7.9% (n = 11), 세 개 이상을 포함하는 균주는 1.4% (n = 2)였다. 두개의 병원성 유전자가 동시에 검출된 경우, stx1과 ibeA는 4 균주, stx2와 ibeA는 7 균주에서 동시에 검출되었다. 병원성 유전자 3개(stx1, stx2, ibeA)와 4개(stx1, stx2, eaeA, ibeA)를 동시에 보유한 균주도 각각 1 균주씩 존재하였다. A 그룹으로 분류된 균주 중 68.7%의 균주가 병원성 유전자를 갖고 있는 것으로 나타났으며, B1과 D 그룹에서도 각각 68.6%와 54.8% 균주에서 병원성 유전자가 검출되었다. B2 그룹은 총 7 균주 중 6 균주에서 병원성 유전자가 검출되어 85.7%의 검출율을 보였다. 전체적으로 A 그룹 (32.9%)에서 가장 많은 병원성 유전자가 검출되었으며 B1 (17.1%), D (12.1%), B2 (4.3%)의 순서로 분포하고 있었다. 계통분류학적 B2 그룹의 경우 다른 그룹에 비해 병원성이 높은 것으로 알려져 있어 병원성 유전자의 검출 비율이 높을 것으로 예상되었다. 하지만, 최근 유전체 및 전사체 연구를 통해 다양한 병원성 전략과 복잡한 숙주-병원균 상호작용이 있음이 확인되었고, 본 연구에 사용된 유전자 외에 매우 다양한 병원성 유전자가 존재하므로 향후 이들 유전자의 존재 유무를 다른 그룹과 비교하는 연구가 더 필요하다(Hogins et al., 2023; Nowrouzian et al., 2009).
병원성 유전자별로 검출 양상을 살펴보면(Table 5), 시가 독소(Shiga toxin) 생성과 관련된 stx1 또는 stx2 유전자를 보유한 균주는 각각 전체의 12.9%와 35.0%로 나타났다. stx1 유전자의 경우 모든 그룹에 고르게 존재하고 있었으나 stx2 유전자는 A 그룹과 B2 그룹에서 상대적으로 높은 비율로 존재하였다. 시가 독소는 시가 독소 생성 대장균(STEC)에 의해 생성되는 강력한 세포 독소로 혈변과 심각한 후유증인 용혈성 요독 증후군을 유발한다(Kaper et al., 2004; Zaragoza et al., 2016). 일반적으로 stx2 유전자에 의해 생성되는 시가 독소 2형(Stx2)은 stx1에 의해 생성되는 시가 독소 1형(Stx1) 보다 높은 독성을 보이는 것으로 알려져 있다(Melton-Celsa, 2014; Zhang et al., 2025).
분리된 대장균 중 29.3%는 신생아 수막염 유발 대장균(NMEC) 및 조류 병원성 대장균(APEC)에서 병원성 인자로 알려진 ibeA 유전자를 보유하고 있는 것으로 나타났다. 뇌 내피 침윤 단백질 A (IbeA)를 코딩하는 ibeA 유전자는 신생아 수막염을 유발하는 대장균(NMEC) 분리주에서 최초로 확인되었으며, 조류 병원성 대장균(APEC)의 고병원성 균주의 26%가 ibeA 양성으로 보고되었다(Cieza et al., 2015; Germon et al., 2005).
또한, 인간에게 고병원성으로 간주되는 STEC 균주의 일부는 장 점막에 부착 및 소실 병변을 유발할 수 있는데, 이때 상피 세포와 긴밀한 부착에 필요한 외막 단백질인 인티민(intimin)을 코딩하는 eaeA 유전자와 세포 손상을 유발하는 장용혈소를 코딩하는 hlyA 유전자와 같은 다양한 요인이 STEC의 독성에 기여한다(Makhado et al., 2022; Paton and Paton, 1998; Paton and Paton, 2002). 연구지역에서 분리된 대장균 중 eaeA 유전자는 1 균주에서 발견되었고, hlyA를 포함하는 균주는 발견되지 않았다.
출혈성 대장염과 혈변을 유발할 수 있는 장출혈성 대장균(EHEC)는 시가 독소 생성 대장균의 아형으로 대표적인 혈청형으로 O157:H7, O26, O121, O103, O111, O145가 알려져 있다(Denamur et al., 2021; Makhado et al., 2022). 특히 O157:H7과 O111 균주는 지난 20년 동안 전세계적으로 많은 식품 매개 질병을 유발했다(Gobin et al., 2018; González-Escalona and Kase, 2019; Schlosserová et al., 2024; Sharapov et al., 2016; Watahiki et al., 2014). 이러한 두 균주를 구별할 수 있는 rfbE와 ORF3.4 유전자는 분리된 대장균에서 검출되지 않았다.
전체적으로 시가 독소 생성 관련 유전자인 stx1과 stx2, 세포 내 침윤 관련 유전자 ibeA가 가장 많이 발견되었으며, 이들 모두 인체 감염 시 심각한 증상을 유발할 수 있으므로 지하수 사용 시 주의가 요구된다.
3.5. 분리 대장균의 병원형 유형
대장균은 전 세계적으로 가장 많이 연구된 미생물로 편리공생, 위장관 질환 또는 장외 병리 등 인간 건강에 미치는 영향으로 인해 매우 다양한 병원형으로 구분된다(Geurtsen et al., 2022; Sarowska et al., 2019). 병원성 대장균은 병원성 인자와 임상 증상에 따라 크게 장내 병원성 대장균(InPEC)과 장외 병원성 대장균(ExPEC)로 분류될 수 있다(Denamur et al., 2021; Geurtsen et al., 2022; Kaper et al., 2004). 최근 광범위한 유전체 분석을 통해 다양한 대장균 병원형이 갖는 병독성 인자의 유전자 정보를 확인하고 이를 활용한 신속하고 정확한 검출 방법이 개발되고 있다(Rathore et al., 2024). 따라서, 일반적인 검사와 더불어 분자생물학적으로 검출된 병독성 유전자의 조합에 따라 대장균의 병원형을 빠르게 판별함으로써 효과적인 치료가 가능함과 동시에 적절한 대응책 마련을 위한 정보를 제공할 수 있다(Rathore et al., 2024).
노성천 유역 지하수에서 분리된 대장균에서 검출된 병원성 유전자에 따라 대장균 병원성 유형으로 분류하였을 때, 전체 분리 대장균 균주의 45.7% (n = 64)가 stx1과 stx2 유전자가 각각 또는 동시에 검출되었을 때 분류되는 시가 독소 생성 대장균(STEC, shiga toxin producing E. coli)로 판별되었으며, ibeA 유전자가 존재할 때 분류되는 ExPEC (extraintestinal pathogenic E. coli)가 20.0% (n = 28)로 분류되었다(Table 5). 또한, stx1과 stx2가 각각 또는 같이 존재하며 동시에 eaeA 유전자가 같이 검출될 때 분류되는 장출혈성 대장균(EHEC, enterohemorrhagic E. coli)은 0.7% (n = 1)로 나타났다.
시가 독소 생성 대장균(STEC)은 인간의 위장관 질환의 주요 원인으로 이들에 의한 감염 증상은 매우 다양하며 특히, 출혈성 대장염(hemorrhagic colitis), 용혈성 요독 증후군(hemolytic-uremic syndrome, UHS) 및 급성 신장 기능 장애로 이어지는 생명을 위협하는 혈전성 미세혈관병증(thrombotic microangiopathy)을 초래할 수 있다(Paton and Paton, 1998). 이들 중 일부는 인체 병원성으로 인해 장출혈성 대장균(EHEC, enterohemorrhagic E. coli)으로 구분되는데, STEC의 병원성 인자인 시가 독소(Stx)과 더불어 장 내벽 세포에 부착할 수 있도록 하는 접착 단백질인 intimin 생성 관련 유전자인 eaeA를 포함한다. 이러한 부착은 EHEC 감염 발병 기전에서 핵심단계로 염증과 설사를 유발한다. EHEC에 의한 감염은 전 세계적으로 빈번하게 발생하고 있으며, 국내에서도 최근 유치원에서 EHEC O157:H7에 의한 대규모 감염도 발생하는 등 지속적인 발병 사례가 보고되고 있다(Heo et al., 2023; Sharapov et al., 2016; Watahiki et al., 2014).
장외 병원성 대장균(ExPEC)은 장내 병원성 대장균(InPEC)와 더불어 인간에게 가장 흔한 병원성 대장균으로, 아형 중 하나인 신생아 뇌수막염 대장균(NMEC, neonatal meningitis E. coli)은 요로 감염을 통해 균혈증을 유발하고 신생아 수막염의 주요 원인이 되기도 한다(Geurtsen et al., 2022; Kaper et al., 2004; Poolman and Wacker, 2016). 또다른 장외 병원성 대장균의 아형인 조류 병원성 대장균(APEC, avian pathogenic E. coli)은 닭, 오리 등을 포함한 가금류에서 다양한 국소 및 전신 감염을 유발 심낭염, 간주위염, 제대염, 패혈증 등 다양한 증상을 유발한다(Mageiros et al., 2021; Sarowska et al., 2019). 이러한 NMEC와 APEC 모두 숙주 조직에 침투하기 위한 침윤 단백질 Ibe를 생성에 관여하는 ibeA 유전자를 내재하고 있으며, 최근 연구를 통해 두 유형이 계통분류학적으로 매우 유사하고 동일한 병원성 유전자를 공유하기 때문에 인간에게 건강 위험을 초래할 가능성이 있다고 보고하였다(Sarowska et al., 2019).
전체적으로 노성천 유역 지하수에서 분리된 대장균의 45.7%가 시가 독소 생성 대장균으로 구분되었고 20.0%는 NMEC와 APEC를 포함하는 ExPEC로 분류되었다. 다만 NMEC와 APEC를 구분하기 위해서는 병원형 구분을 위한 특이적인 병원성 유전자 검출 연구가 추가로 수행되어야 할 것으로 사료된다. EHEC로 분류된 균주는 심각한 감염 증상을 일으키는 O157이나 O111 특이적인 유전자가 검출되지 않았기 때문에 다른 혈청형 일 것으로 판단된다.
단일 또는 여러 병원성 유전자를 소유한다고 해도 해당 균주가 특정 숙주에서 질병을 유발하는 데 적합한 병원성 유전자 조합을 갖고 있지 않는 한 반드시 병원성이라는 것을 나타내는 것은 아니다(Masters et al., 2011). 하지만 병원균간 항생제 내성 및 이동성 병원성 인자의 빠른 전이가 발생할 가능성 또한 존재하기 때문에 농촌지역 지하수의 공중 보건을 보호하기 위해 대장균에 대한 모니터링 강화가 필요할 것으로 판단된다.
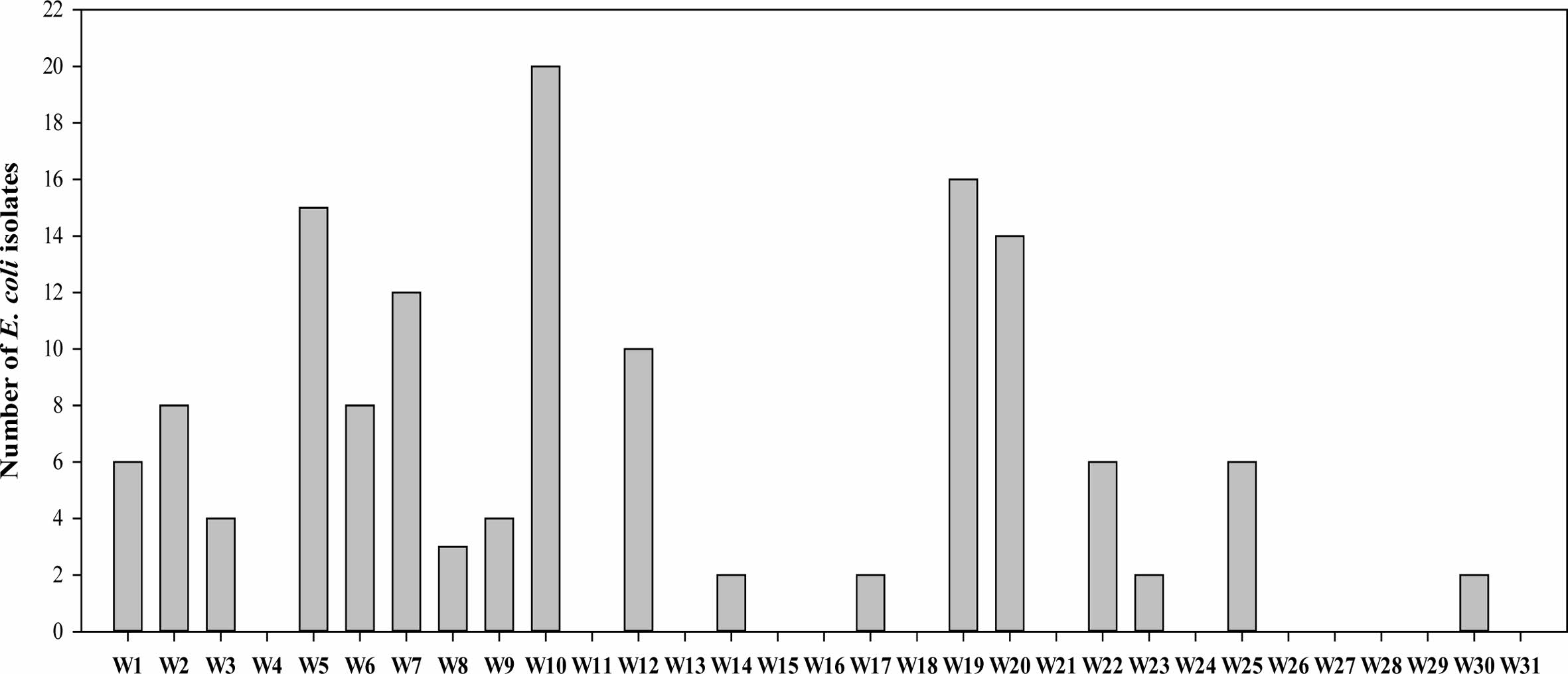
|
Fig. 2 Spatial variation of isolated E. coli numbers from groundwater samples. |
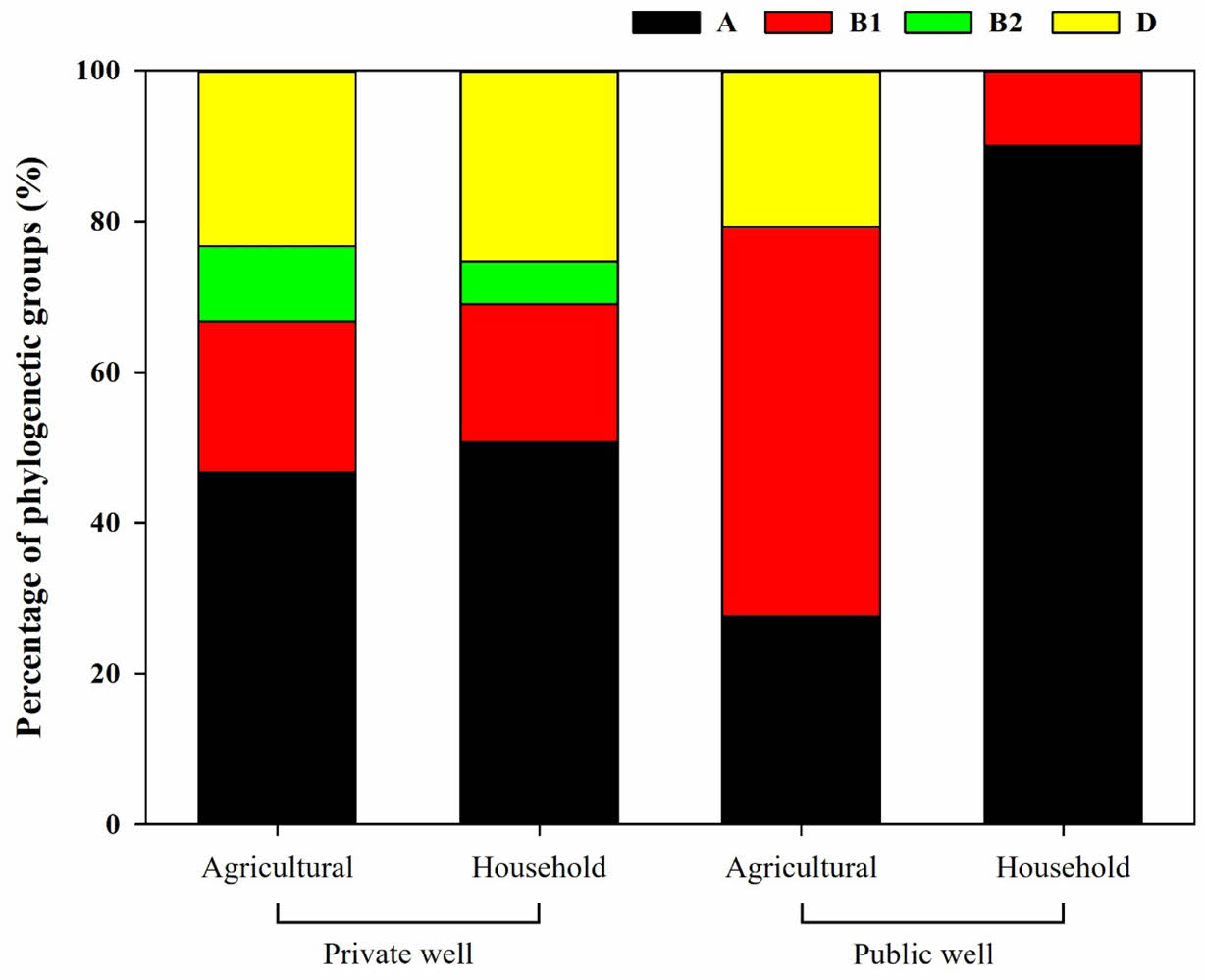
|
Fig. 3 Distribution of phylogenetic groups of E. coli isolated from groundwater samples. |
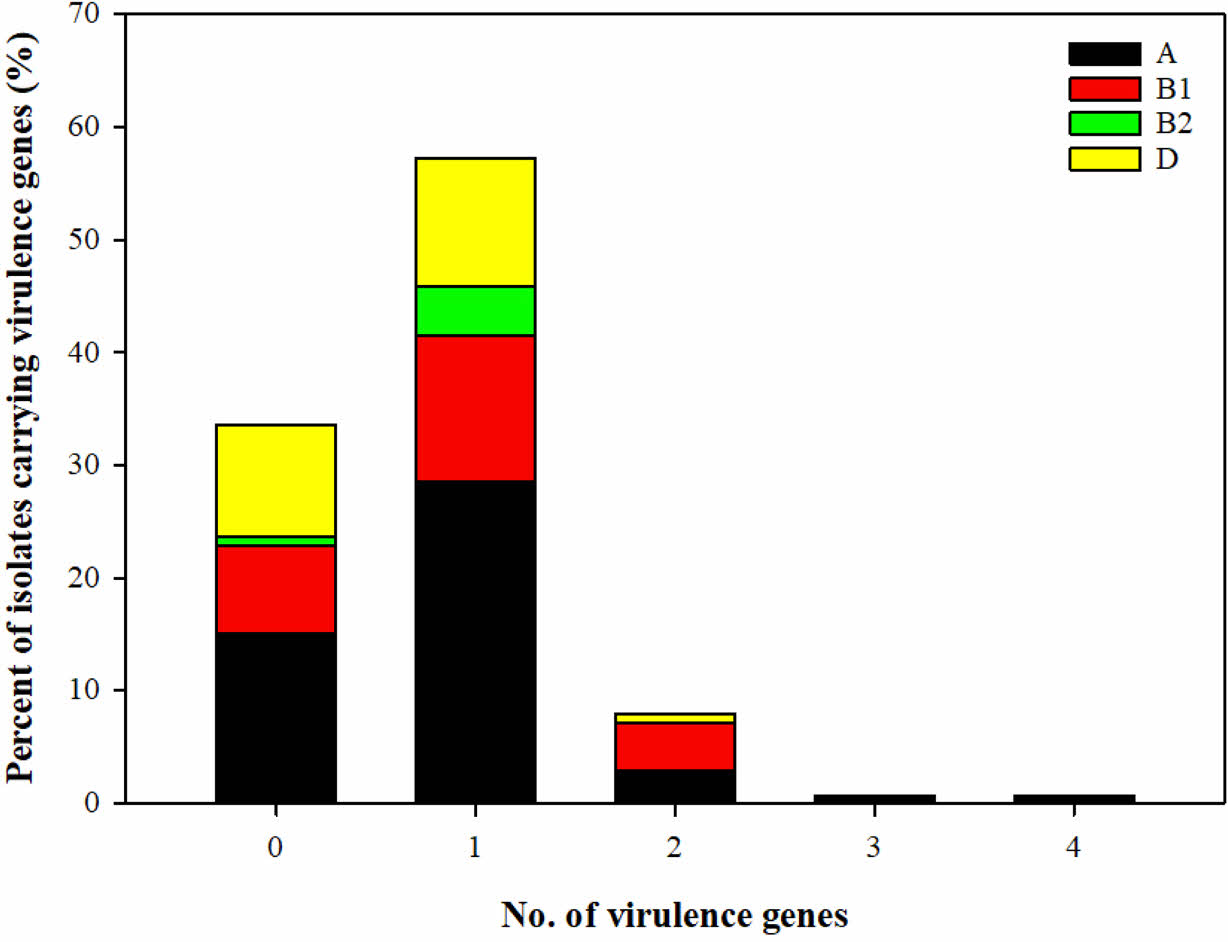
|
Fig. 4 Comparative distribution of the number of virulence genes carried by individual E. coli isolates. |
|
Table 2 Summary statistics of measured parameters of groundwater samples |
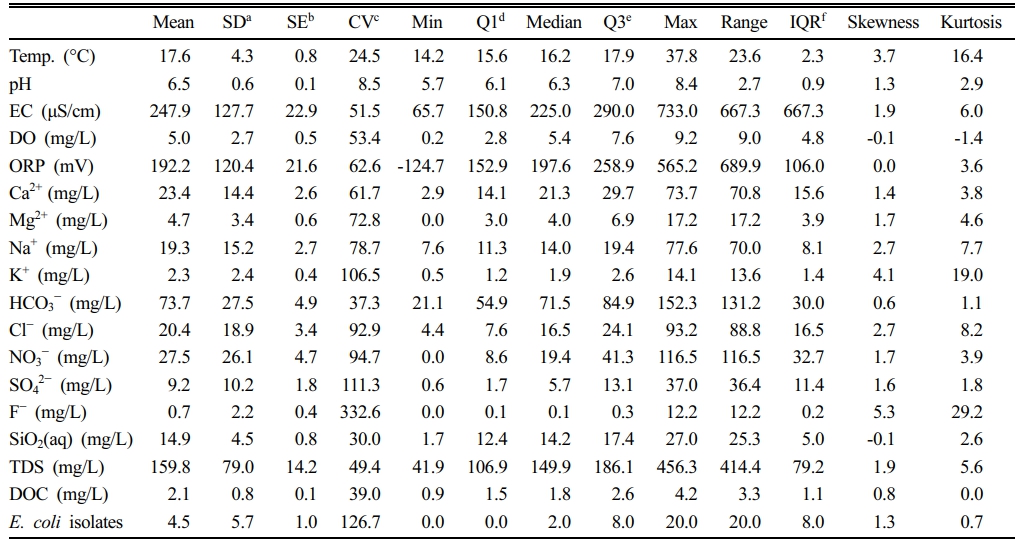
Notes: Number of samples n = 31, aSD: standard deviation, bSE: standard error, cCV: coefficient of variation, dQ1: first Quartile, eQ3: third Quartile, fIQR: inter Quartile Range. |
|
Table 5 Occurrence of virulence genes and pathotypes in E. coli isolates |
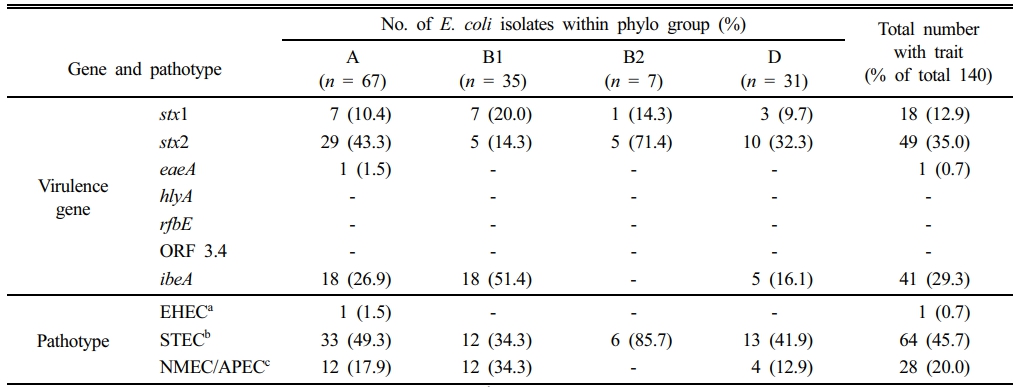
aEHEC (entrohemorrhagic E. coli): stx1 or/and stx2, eaeA present, bSTEC (shiga toxin producing E. coli): stx1 or/and stx2 present, cNMEC/APEC (potentially neonatal meningitis E. coli / avian pathogenic E. coli): ibeA present |
본 연구는 전형적인 농촌지역인 노성천 유역의 지하수에서 대장균을 분리하고 이들의 계통분류학적 및 병원성 특성을 분석하였다. 연구지역의 지하수는 대부분 생활용 및 농업용으로 사용되는 천부대수층으로 지표수의 영향을 크게 받고 있는 것으로 보인다. 지하수 시료로부터 140 균주의 대장균을 분리하였으며 개인용 관정에서 가장 많은 대장균이 분리되었다. 계통분류학적 분석 결과 대부분 A 그룹과 B1, D 그룹으로 구성되어 있었으며 병원성이 높은 B2 그룹은 상대적으로 적게 존재하였다. 분리된 대장균 중 66.4%에서 하나 이상의 병원성 유전자가 존재하는 것으로 나타났으며, 9.3%의 균주에서는 두 개 이상의 병원성 유전자가 동시에 검출되었다. 병원성 유전자 중 시가 독소 생성 관련 유전자인 stx1과 stx2가 가장 많이 검출되고 다음으로 세포 침윤과 관련된 ibeA 유전자가 많이 존재하였다. 검출된 병원성 유전자를 바탕으로 병원형으로 분류하였을 때, 대부분 시가 독소 생성 대장균(STEC) 및 신생아 수막염 대장균(NMEC)과 조류 병원성 대장균(APEC)을 포함하는 장외 병원성 대장균(ExPEC)으로 구분되었다. 비록 NMEC와 APEC가 정확히 구분되지는 않지만 두 부류 모두 인간 감염의 가능성이 존재하므로 주의를 기울일 필요가 있다.
연구지역의 지하수 시료에 대장균이 광범위하게 존재하고 있으며 기타 병원성균들도 다수 검출되는 특징으로 보아, 해당 관정의 설치 또는 관리가 미흡한 것으로 판단된다(Kim et al., 2022). 농촌 지역의 경우 축산 분뇨 사용과 개인 하수의 부적절한 처리 등으로 인한 지하수 오염이 증가하는 추세이다. 오염된 지하수는 농작물 수확량 감소 및 품질 저하를 일으킬 수 있으며, 대장균으로 오염된 지하수를 음용하거나 식품 등의 세척에 사용할 경우 감염 가능성이 현저히 증가할 수 있다. 따라서, 연구지역 뿐 만 아니라 지하수를 음용수나 생활용수로 사용하지만 관리가 미흡한 지역에서 안전한 지하수 사용을 위해 주기적인 관리가 필요하다.
본 연구는 과학기술정보통신부에서 지원한 한국지질자원연구원 기본사업(GP2025-011)의 도움으로 수행되었습니다.
- 1. Abbas, N., Song, S., Chang, M.-S., and Chun, M.-S., 2023, Point-of-care diagnostic devices for detection of Escherichia coli O157:H7 using microfluidic systems: A focused review, Biosensors, 13(7), 741.
-

- 2. Abia, A.L., Schaefer, L., Ubomba-Jaswa, E., and Le Roux, W., 2017, Abundance of pathogenic Escherichia coli virulence-associated genes in well and borehole water used for domestic purposes in a peri-urban community of South Africa, Int. J. Environ. Res. Public Health, 14(3), 320.
-

- 3. Agin, T.S. and Wolf, M.K., 1997, Identification of a family of intimins common to Escherichia coli causing attaching-effacing lesions in rabbits, humans, and swine, Infect. Immun., 65(1), 320-326.
-

- 4. Alhadlaq, M.A., Aljurayyad, O.I., Almansour, Ayidh, Al-Akeel, S.I, Alzahrani, K.O., Alsalman, S.A., Yahya, R., Al-Hindi, R.R., Hakami, M.A., Alshahrani, S.D., Alhumeed, N.A., Al Moneea, A.M, Al-Seghayer, M.S., AlHarbi, A.L., Al-Reshoodi, F.M., and Alajel, S., 2024, Overview of pathogenic Escherichia coli, with a focus on Shiga toxin-producing serotypes, global outbreaks (1982-2024) and food safety criteria, Gut Pathog., 16(1), 57.
-

- 5. Bagordo, F., Brigida, S., Grassi, T., Caputo, M.C., Apollonio, F., Carlo, L.D., Savino, A.F., Triggiano, F., Turturro, A.C., Donno, A.D., Montagna, M.T., and Giglio, O.D., 2024, Factors influencing microbial contamination of groundwater: A systematic review of field-scale studies, Microorganisms, 12(5), 913.
-

- 6. Bartzas, G., Tinivella, F., Medini, L., Zaharaki, D., and Komnitsas, K., 2015, Assessment of groundwater contamination risk in an agricultural area in north Italy, Inf. Process. Agric., 2(2), 109-129.
-

- 7. Bastin, D.A. and Reeves, P.R., 1995, Sequence and analysis of the O antigen gene (rfb) cluster of Escherichia coli 0111, Gene, 164(1), 17-23.
-

- 8. Bilge, S.S., Vary, J.C., Jr., Dowell, S.F., and Tarr, P.I., 1996, Role of the Escherichia coli O157:H7 O side chain in adherence and analysis of an rfb locus, Infect. Immun., 64(11), 4795-4801.
-

- 9. Boulding, J.R. and Ginn, J.S., 2016. Practical handbook of soil, vadose zone, and ground-water contamination: assessment, prevention, and remediation. CRC Press.
-

- 10. Chern, E.C., Siefring, S., Paar, J., Doolittle, M., and Haugland, R.A., 2011, Comparison of quantitative PCR assays for Escherichia coli targeting ribosomal RNA and single copy genes, Lett. Appl. Microbiol., 52(3), 298-306.
-

- 11. Cho, B.-W., Yun, U., Lee, B.-D., and Ko, K.-S., 2012, Hydrogeological characteristics of the Wangjeon-ri PCWC area, Nonsan-city, with an emphasis on water level variations, The J. Eng. Geol., 22(2), 195-205.
-

- 12. Cieza, R.J., Hu, J., Ross, B.N., Sbrana, E., and Torres, A.G., 2015, The IbeA invasin of adherent-invasive Escherichia coli mediates interaction with intestinal epithelia and macrophages, Infect. Immun., 83(5), 1904-1918.
-

- 13. Clermont, O., Bonacorsi, S., and Bingen, E., 2000, Rapid and simple determination of the Escherichia coli phylogenetic group, Appl. Environ. Microbiol., 66(10), 4555-4558.
-

- 14. Clermont, O., Christenson, J.K., Denamur, E., and Gordon, D.M., 2013, The clermont Escherichia coli phylo-typing method revisited: improvement of specificity and detection of new phylo-groups, Environ. Microbiol. Rep., 5(1), 58-65.
-

- 15. Denamur, E., Clermont, O., Bonacorsi, S., and Gordon, D., 2021, The population genetics of pathogenic Escherichia coli, Nat. Rev. Microbiol., 19(1), 37-54.
-

- 16. Dombek, P.E., Johnson, L.K., Zimmerley, S.T., and Sadowsky, M.J., 2000, Use of repetitive DNA sequences and the PCR to differentiate Escherichia coli isolates from human and animal sources, Appl. Environ. Microbiol., 66(6), 2572-2577.
-

- 17. Escobar-Páramo, P., Menac'h, A.L., Gall, T.L., Amorin, C., Gouriou, S., Picard, B., Skurnik, D., and Denamur, E., 2006, Identification of forces shaping the commensal Escherichia coli genetic structure by comparing animal and human isolates, Environ. Microbiol., 8(11), 1975-1984.
-

- 18. Gelting, R.J., Baloch, M.A., Zarate-Bermudez, M.A., and Selman, C., 2011, Irrigation water issues potentially related to the 2006 multistate E. coli O157:H7 outbreak associated with spinach, Agric. Water Manag., 98(9), 1395-1402.
-

- 19. Germon, P., Chen, Y.-H., He, L., Blanco, J.E., Brée, A., Schouler, C., Huang, S.-H., and Moulin-Schouleur, M., 2005, ibeA, a virulence factor of avian pathogenic Escherichia coli, Microbiol., 151(4), 1179-1186.
-

- 20. Geurtsen, J., Been, M.d., Weerdenburg, E., Zomer, A., McNally, A., and Poolman, J., 2022, Genomics and pathotypes of the many faces of Escherichia coli, FEMS Microbiol. Rev., 46(6), fuac031.
-

- 21. Gilmore, M.S. and Ferretti, J.J., 2003, The thin line between gut commensal and pathogen, Sci., 299(5615), 1999-2002.
-

- 22. Gobin, M., Hawker, J., Cleary, P., Inns, T., Gardiner, D., Mikhail, A., McCormick, J., Elson, R., Ready, D., Dallman, T., Roddick, I., Hall, I., Willis, C., Crook, P., Godbole, G., Tubin-Delic, D., and Oliver, I., 2018, National outbreak of Shiga toxin-producing Escherichia coli O157:H7 linked to mixed salad leaves, United Kingdom, 2016, Euro Surveill., 23(18), 17-00197.
-

- 23. González-Escalona, N. and Kase, J.A., 2019, Virulence gene profiles and phylogeny of Shiga toxin-positive Escherichia coli strains isolated from FDA regulated foods during 2010-2017, PLoS One, 14(4), e0214620.
-

- 24. Govindarajan, D.K., Eskeziyaw, B.M., Kandaswamy, K., and Mengistu, D.Y., 2024, Diagnosis of extraintestinal pathogenic Escherichia coli pathogenesis in urinary tract infection, Curr. Res. Microb. Sci., 7, 100296.
-

- 25. Green, M.R. and Sambrook, J., 2012. Molecular cloning: A Laboratory Manual, 4th., 448. Cold Spring Harbor Laboratory Press.
- 26. Gwimbi, P., George, M., and Ramphalile, M., 2019, Bacterial contamination of drinking water sources in rural villages of Mohale Basin, Lesotho: exposures through neighbourhood sanitation and hygiene practices, Environ. Health. Prev. Med., 24(1), 33.
-

- 27. Heo, N., Lee, J., Kim, Y., Lee, D., Heo, S.J., Park, Y.S., Yun, J.W., and Kim, Y.C., 2023, The epidemiological and clinical characteristics of the largest outbreak of enterohemorrhagic Escherichia coli in Korea, J. Korean Med. Sci., 38(15), e117.
-

- 28. Hogins, J., Xuan, Z., Zimmern, P.E., and Reitzer, L., 2023, The distinct transcriptome of virulence-associated phylogenetic group B2 Escherichia coli, Microbiol. Spectr., 11(5), e02085-02023.
-

- 29. Holvoet, K., Sampers, I., Seynnaeve, M., and Uyttendaele, M., 2014, Relationships among hygiene indicators and enteric pathogens in irrigation water, soil and lettuce and the impact of climatic conditions on contamination in the lettuce primary production, Int. J. Food Microbiol., 171, 21-31.
-

- 30. Jang, J., Di, D.Y.W., Lee, A., Unno, T., Sadowsky, M.J., and Hur, H.-G., 2014, Seasonal and genotypic changes in Escherichia coli phylogenetic groups in the Yeongsan River basin of South Korea, PLoS One, 9(7), e100585.
-

- 31. Jang, J., Hur, H.‐G., Sadowsky, M.J., Byappanahalli, M.N., Yan, T., and Ishii, S., 2017, Environmental Escherichia coli: ecology and public health implications-a review, J. Appl. Microbiol., 123(3), 570-581.
-

- 32. Jang, J., Unno, T., Lee, S.W., Cho, K.H., Sadowsky, M.J., Ko, G., Kim, J.H., and Hur, H.-G., 2011, Prevalence of season‐specific Escherichia coli strains in the Yeongsan River Basin of South Korea, Environ. Microbiol., 13(12), 3103-3113.
-

- 33. Jay, M.T., Cooley, M., Carychao, D., Wiscomb, G.W., Sweitzer, R.A., Crawford-Miksza, L., Farrar, J.A., Lau, D.K., O'Connell, J., Millington, A., Asmundson, R.V., Atwill, E.R., and Mandrell, R.E., 2007, Escherichia coli O157:H7 in feral swine near spinach fields and cattle, central California coast, Emerg. Infect. Dis., 13(12), 1908-1911.
-

- 34. Jha, M.K., Shekhar, A., and Jenifer, M.A., 2020, Assessing groundwater quality for drinking water supply using hybrid fuzzy-GIS-based water quality index, Water Res., 179, 115867.
-

- 35. Kaper, J.B., Nataro, J.P., and Mobley, H.L.T., 2004, Pathogenic Escherichia coli, Nat. Rev. Microbiol., 2(2), 123-140.
-

- 36. Kim, D.-H., Oh, Y.H., Lee, B.-J., and Lee, J.-Y., 2022, Hydrogeochemistry and microbial community structure of groundwater in an agricultural area, J. Soil Groundwater Environ., 27(2), 61-75.
- 37. KOSIS, 2025. https://kosis.kr [accessed 25.06.02].
- 38. Kwon, H.-I., Koh, D.-C., Jung, Y.-Y., Kim, D.-H., and Ha, K., 2020, Evaluating the impacts of intense seasonal groundwater pumping on stream–aquifer interactions in agricultural riparian zones using a multi-parameter approach, J. Hydrol., 584, 124683.
-

- 39. Lee, D. and Murphy, H.M., 2020, Private wells and rural health: Groundwater contaminants of emerging concern, Curr. Environ. Health Rep., 7(2), 129-139.
-

- 40. Li, D., Sharp, J.O., Saikaly, P.E., Ali, S., Alidina, M., Alarawi, M.S., Keller, S., Hoppe-Jones, C., and Drewes, J.E., 2012, Dissolved organic carbon influences microbial community composition and diversity in managed aquifer recharge systems, Appl. Environ. Microbiol., 78(19), 6819-6828.
-

- 41. Mageiros, L., Méric, G., Bayliss, S.C., Pensar, J., Pascoe, B., Mourkas, E., Calland, J.K., Yahara, K., Murray, S., Wilkinson, T.S., Williams, L.K., Hitchings, M.D., Porter, J., Kemmett, K., Feil, E.J., Jolley, K.A., Williams, N.J., Corander, J., and Sheppard, S.K., 2021, Genome evolution and the emergence of pathogenicity in avian Escherichia coli, Nat. Commun., 12(1), 765.
-

- 42. Majowicz, S.E., Scallan, E., Jones-Bitton, A., Sargeant J.M., Stapleton, J., Angulo, F.J., Yeung, D.H., and Kirk, M.D., 2014, Global incidence of human shiga toxin–producing Escherichia coli infections and deaths: A systematic review and knowledge synthesis, Foodborne Pathog. Dis., 11(6), 447-455.
-

- 43. Makhado, U.G., Foka, F.E.T., Tchatchouang, C.-D.K., Ateba, C.N., and Manganyi, M.C., 2022, Detection of virulence gene of Shiga toxin-producing Escherichia coli (STEC) strains from animals with diarrhoea and water samples in the North-West Province, South Africa, Gene Rep., 27, 101617.
-

- 44. Martins, M.T., Rivera, G., Clark, D.L., Stewart, M.H., Wolfe, R.L., and Olson, B.H., 1993, Distribution of uidA gene sequences in Escherichia coli isolates in water sources and comparison with the expression of beta-glucuronidase activity in 4-methylumbelliferyl-beta-D-glucuronide media, Appl. Environ. Microbiol., 59(7), 2271-2276.
-

- 45. Masters, N., Wiegand, A., Ahmed, W., and Katouli, M., 2011, Escherichia coli virulence genes profile of surface waters as an indicator of water quality, Water Res., 45(19), 6321-6333.
-

- 46. Melton-Celsa, A.R., 2014, Shiga toxin (Stx) classification, structure, and function, Microbiol. Spectr., 2(4), 10.1128/microbiolspec.ehec-0024-2013.
-

- 47. Mueller, D.K. and Helsel, D.R., 1996. Nutrients in the nation's waters: too much of a good thing?, 1136. US Geol. Surv. Circular.
-

- 48. Mutileni, N., Mudau, M., and Edokpayi, J.N., 2023, Water quality, geochemistry and human health risk of groundwater in the Vyeboom region, Limpopo province, South Africa, Sci. Rep., 13(1), 19071.
-

- 49. Nowrouzian, F.L., Östblom, A.E., Wold, A.E., and Adlerberth, I., 2009, Phylogenetic group B2 Escherichia coli strains from the bowel microbiota of Pakistani infants carry few virulence genes and lack the capacity for long-term persistence, Clin. Microbiol. Infect., 15(5), 466-472.
-

- 50. Odonkor, S.T. and Mahami, T., 2020, Escherichia coli as a tool for disease risk assessment of drinking water sources, Int. J. Microbiol., 2020(1), 2534130.
-

- 51. Ohad, S., Vaizel-Ohayon, D., Rom, M., Guttman, J., Berger, D., Kravitz, V., Pilo, S., Huberman, Z., Kashi, Y., and Rorman, E. 2015, Microbial source tracking in adjacent karst springs, Appl. Environ. Microbiol., 81(15), 5037-5047.
-

- 52. Pagadala, S., Marine, S.C., Micallef, S.A., Wang, F., Pahl, D.M., Melendez, M.V., Kline, W.L., Oni, R.A., Walsh, C.S., Everts, K.L., and Buchanan, R.L., 2015, Assessment of region, farming system, irrigation source and sampling time as food safety risk factors for tomatoes, Int. J. Food Microbiol., 196, 98-108.
-

- 53. Park, J., Kim, J.S., Kim, S., Shin, E., Oh, K.-H., Kim, Y., Kim, C.H., Hwang, M.A., Jin, C.M., Na, K., Lee, J., Cho, E., Kang, B.-H., Kwak, H.-S., Seong, W.K., and Kim, J., 2018, A waterborne outbreak of multiple diarrhoeagenic Escherichia coli infections associated with drinking water at a school camp, Int. J. Infect. Dis., 66, 45-50.
-

- 54. Paton, A.W. and Paton, J.C., 1998, Detection and characterization of shiga toxigenic Escherichia coli by using multiplex PCR assays for stx1, stx2, eaeA, enterohemorrhagic E. coli hlyA, rfb O111, and rfb O157, J. Clin. Microbiol., 36(2), 598-602.
-

- 55. Paton, A.W. and Paton, J.C., 2002, Direct detection and characterization of shiga toxigenic Escherichia coli by multiplex PCR for stx1, stx2, eae, ehxA, and saa, J. Clin. Microbiol., 40(1), 271-274.
-

- 56. Poolman, J.T. and Wacker, M., 2016, Extraintestinal pathogenic Escherichia coli, a common human pathogen: Challenges for vaccine development and progress in the field, J. Infect. Dis., 213(1), 6-13.
-

- 57. Rathore, P., Basnet, A., Kilonzo-Nthenge, A., Dumenyo, K., Yadegari, Z., and Taheri, A., 2024, Rapid detection of pathogenic E. coli based on CRISPR Cas system, Front. Microbiol., 15, 1423478.
-

- 58. Rojas-Lopez, M., Monterio, R., Pizza, M., Desvaux, M., and Rosini, R., 2018, Intestinal pathogenic Escherichia coli: Insights for vaccine development, Front. Microbiol., 9, 440.
-

- 59. Sarowska, J., Futoma-Koloch, B., Jama-Kmiecik, A., Frej-Madrzak, M., Ksiazczyk, M., Bugla-Ploskonska, G., and Choroszy-Krol, I., 2019, Virulence factors, prevalence and potential transmission of extraintestinal pathogenic Escherichia coli isolated from different sources: recent reports, Gut Pathog., 11(1), 10.
-

- 60. Schlosserová, K., Daniel, O, Labská, K., Jakubů, V., Stárková, T., Bílý, J., Dresler, J., Lang, C., Fruth, A., Flieger, A., Žemličková, H., Bielaszewska, M., and Havlíčková, M., 2024, Enteroaggregative Escherichia coli: Frequent, yet underdiagnosed pathotype among E. coli O111 strains isolated from children with gastrointestinal disorders in the Czech Republic, Int. J. Med. Microbiol., 316, 151628.
-

- 61. Sharapov, U.M., Wendel, A.M., Davis, J.P., Keene, W.E., Farrar, J., Sodha, S., Hyytia-Trees, E., Leeper, M., Gerner-Smidt, P., Griffin, P.M., Braden, C., and TEAM, O.I., 2016, Multistate outbreak of Escherichia coli O157:H7 infections associated with consumption of fresh spinach: United States, 2006, J. Food Prot., 79(12), 2024-2030.
-

- 62. Shimizu, T., Ohta, Y., and Noda, M., 2009, Shiga toxin 2 is specifically released from bacterial cells by two different mechanisms, Ifect. Immun., 77(7), 2813-2823.
-

- 63. Stoppe, N.d.C., Silva, J.S., Carlos, C., Sato, M.I.Z., Saraiva, A.M., Ottoboni, L.M.M., and Torres, T.T., 2017, Worldwide phylogenetic group patterns of Escherichia coli from commensal human and wastewater treatment plant isolates, Front. Microbiol., Volume 8 - 2017.
-

- 64. Tousi, E.G., Duan, J.G., Gundy, P.M., Bright, K.R., and Gerba, C.P., 2021, Evaluation of E. coli in sediment for assessing irrigation water quality using machine learning, Sci. Total Environ., 799, 149286.
-

- 65. Unno, T., Han, D., Jang, J., Lee, S.-N., Ko, G.P., Choi, H.Y., Kim, J.H., Sadowsky, M.J., and Hur, H.-G., 2009, Absence of Escherichia coli phylogenetic group B2 strains in humans and domesticated animals from Jeonnam Province, Republic of Korea, Appl. Environ. Microbiol., 75(17), 5659-5666.
-

- 66. USEPA, 2014. Method 1603: Escherichia coli (E. coli) in water by membrane filteration using modified membrane-thermotolerant Escherichia coli agar (modified mTEC). EPA-821-R-14-010, U.S. Environmental Protection Agency.
- 67. Viazis, S., Bazaco, M.C., Blessington, T., Federman, S.L.S., Lindsay, M., Minor, T., Nork, B., Mattioli, M.C., Marshall, K.E., Wise, M.E., Weed, B., Haendiges, J., Gieraltowski, L., and Nolte, K., 2025, An overview of farm investigation findings associated with outbreaks of shiga toxin-producing Escherichia coli infections linked to leafy greens: 2009-2021, J. Food Prot., 88(7), 100542.
-

- 68. Wang, S., Niu, C., Shi, Z., Xia, Y., Yaqoob, M., Dai, J., and Lu, C., 2011, Effects of ibeA deletion on virulence and biofilm formation of avian pathogenic Escherichia coli, Infect. Immun., 79(1), 279-287.
-

- 69. Watahiki, M., Isobe, J., Kimata, K., Shima, T., Kanatani, J.-I., Shimizu, M., and Nagata, A., 2014, Characterization of enterohemorrhagic Escherichia coli O111 and O157 strains isolated from outbreak patients in Japan, J. Clin. Microbiol., 52(8), 2757-2763.
-

- 70. Xue, X. and Zhang, Y., 2025, Review of the detection of pathogenic Escherichia coli based-microchip technology, Anal. Sci., 41(3), 225-236.
-

- 71. Yang, Y., Yan, X., Redding, M., Gu, G., Luo, Y., Patel, J., and Nou, X., 2025, Biofilm formation of Escherichia coli O157:H7 strains associated with recent reoccurring lettuce outbreaks, Food Microbiol., 128, 104728.
-

- 72. Yoon, J., Park, S., Choi, H., Kim, D.H., Kim, M., Yun, S.-T., Kim, Y., and Kim, H.-K., 2020, Analysis of groundwater quality and contamination factors in livestock region, South Korea, J. Soil Groundwater Environ., 25(4), 98-105.
- 73. Zaragoza, W.J., Teplitski, M., and Fagerquist, C.K., 2016, Shiga toxin-producing Escherichia coli: Detection, differentiation, and implications for food safety, EDIS, 2016(5), 6.
-

- 74. Zhang, H., Yamamoto, E., Markell, A., Carrillo, C., and Locas, A., 2025, Prevalence of shiga toxin-producing Escherichia coli (STEC) and risk characterization based on virulence genes in retail raw ground meat of beef, veal, and lamb in Canada, J. Food Prot., 88(5), 100483.
-

 This Article
This Article
-
2025; 30(4): 53-66
Published on Aug 28, 2025
- 10.7857/JSGE.2025.30.4.053
- Received on Jun 9, 2025
- Revised on Jun 26, 2025
- Accepted on Jul 7, 2025
 Services
Services
Shared
 Correspondence to
Correspondence to
- Dong-Hun Kim
-
Groundwater Environment Research Center, Geo-Environment Research Division, Korea Institute of Geoscience
- E-mail: donghun@kigam.re.kr